고정 헤더 영역
상세 컨텐츠
본문 제목
[KOR] [Pyimpute] Example : A brief introduction to Species Distribution Models in Python (by Daniel Furman)
본문

written in 2022, Jan 23
시중에 나온 많은 서적들이 sklearn을 활용하는 예제를 통해 많은 학생들이 머신러닝을 입문하는 데에 도움을 주고 있지만, 나와 같이 지리정보시스템(GIS)와 결합하여 연구하고자하는 경우 약간의 아쉬움을 갖게 된다.
위 예제에서 사용된 pyimpute 패키지는 머신러닝 패키지인 sklearn과 지리정보시스템과 관련된 레스터 데이터를 다루는 rasterio 패키지를 연결하는 역할을 수행하기 때문에 이 포스팅을 통해 소개해주고 싶다.
해당 예제의 Species Distribution Model(이하 SDM; 종 분포 모델링) 과정에서는 pyimpute 패키지의 지도학습 분류 문제를 풀고, 여러 가지 머신러닝 분류기에 대한 교차 검증 과정을 포함하고 있어 위치정보 데이터 기반 머신러닝 기술을 습득하는 데 많은 도움이 된다.
아래에 첨부되어 있는 유튜브 동영상을 통해 과정을 따라해보고, 설명에 나오지 않은 부분이나 내가 추가적으로 이해한 부분에 대해서 기술하는 점에 중점을 두고 이 포스트를 작성하였다.
Overview
이 튜토리얼에서는 아래 3단계를 따르는 과정으로 진행된다. 파이썬이나 패키지를 다운하는 준비 과정, 데이터 획득 과정 및 QGIS 플러그인을 통한 입력(INPUT) 자료 생성은 내용상 생략하도록 한다.
- 데이터 전처리: 데이터프레임 칼럼 정렬 및 레스터데이터 리스트 생성
- 머신러닝: 데이터 학습 및 모델 평가
- 시각화
Procedure
0. Data Description
JTREE.shp: 조슈아 트리(Joshua tree)의 생존여부, 경도(lon), 위도(lat)를 가지고 있는 벡터 데이터bioclim/bclim*.asc: World Climate 데이터 중 조슈아 연구지역으로 쿠키커팅 처리한 생물 기후 특징 19가지 (연평균 기온, 연간 온도 범위, 연간 강수량 등을 포함하고 있음. 자세한 내용은 참고자료 링크 참조)
0. Key Terms
- Species Suitability : 종 적합성
- Species Distribution Model : 종 분포 모델링
1. Data Processing
JTREE_TRAIN_VEC.csv 파일을 만드는 방법은 크게 두 가지가 있다. 두 방법 모두 같은 결과물을 생성하지만 2번 방법이 조금 더 시간을 단축할 수 있다.
- 깃허브 소스코드의
data-pre-processing.R파일로 샘플링 (by Python-sdm.ipnb) - QGIS Plugins Point sampling tool을 활용하여 샘플링 (by GGRS Youtube)
하지만 JTREE_TRAIN_VEC.csv 파일을 생성하다보면, QGIS 상에서 레이어 순서에 따라 생성된 .csv 파일의 칼럼 순서가 바뀔 수 있는데 이를 고치기 위해서 다음 코드를 사용할 수 있다.
import pandas as pd
>>> train_vec = pd.read_csv("INPUT/JTREE_TRAIN_VEC.csv")
>>> train_vec.head()
>>> train_vec = train_vec[sorted(train_vec.columns)]
>>> train_vec
sorted() 함수를 쓰는 경우 1, 2, ..., 12와 같은 일반적인 순서가 1, 10, 11, 12, 2, 3, ..., 9가 됨에 주의하자.
한편 pyimpute.load_target() 함수의 유일한 인자인 explanatory_rasters에 들어가기 위한 .asc 파일경로 리스트, 다른 표현으로 레스터 파일경로 리스트를 만들기 위해 glob.glob() 함수를 썼다.
(explanatory_rasters : List of Paths to GDAL rasters containing explanatory variables, 소스 코드 참조)
>>> raster_features = sorted(glob.glob('INPUT/bclim*.asc'))
>>> print(raster_features)
['INPUT\\bclim1.asc', 'INPUT\\bclim10.asc', 'INPUT\\bclim11.asc', 'INPUT\\bclim12.asc', 'INPUT\\bclim13.asc', 'INPUT\\bclim14.asc', 'INPUT\\bclim15.asc', 'INPUT\\bclim16.asc', 'INPUT\\bclim17.asc', 'INPUT\\bclim18.asc', 'INPUT\\bclim19.asc', 'INPUT\\bclim2.asc', 'INPUT\\bclim3.asc', 'INPUT\\bclim4.asc', 'INPUT\\bclim5.asc', 'INPUT\\bclim6.asc', 'INPUT\\bclim7.asc', 'INPUT\\bclim8.asc', 'INPUT\\bclim9.asc']
os.listdir()과 glob.glob() 모두 파일 리스트를 가져오지만, 특히나 glob.glob()는 검색시 사용했던 경로명까지 전부 가져오는 장점이 있어 위 코드에서 활용하였다.
2. ML
머신러닝 패키지 sklearn 예제 중 지도학습 분류 문제에서 자주 보듯이, 학습용 데이터(training data)를 반응변수(response var.)와 예측변수(predictor)로 나누어 학습을 진행한다.
단, 이 예제에서 반응변수는 생존여부를 나타낸 'CLASS' 칼럼이 된다. 학습용 데이터의 반응변수 및 예측변수의 개수가 같은지 확인하기 위해서 .shape(attribute)로 확인한다.
import numpy
from pyimpute import load_targets
train_xs, train_y = train_vec.iloc[:, 1:20].values, train_vec.iloc[:, 0].values
target_xs, raster_info = load_targets(raster_features)
train_xs.shape, train_y.shape
이후에는 sklearn.model_selection을 통해서 CLASS_MAP으로 정의된 머신러닝 분류기 딕셔너리에서 하나씩 모델을 꺼내와서 모델 피팅 및 공간 예측을 실행하고 정확도(accuracy)를 기준으로 모델을 평가한다. 여기서는 k-fold 교차검증 기법을 활용하여 특정 훈련 데이터에 의한 과적합을 방지하였다.
>>> from sklearn.ensemble import RandomForestClassifier
>>> from sklearn.ensemble import ExtraTreesClassifier
>>> from xgboost import XGBClassifier
>>> from lightgbm import LGBMClassifier
>>>
>>> CLASS_MAP = {
>>> 'RF': (RandomForestClassifier(random_state=1)),
>>> 'ET': (ExtraTreesClassifier(random_state=1)),
>>> 'XGB': (XGBClassifier(random_state=1)),
>>> 'LGBM': (LGBMClassifier(random_state=1))
>>> }
>>>
>>> from pyimpute import impute
>>> from sklearn import model_selection
>>>
>>> for folder in glob.glob('OUTPUT/*'):
>>> shutil.rmtree(folder)
>>>
>>> for name, (model) in CLASS_MAP.items():
>>> k = 5
>>> kf = model_selection.KFold(n_splits=k)
>>> accuracy_scores = model_selection.cross_val_score(model, train_xs, train_y, cv=kf, scoring='accuracy')
>>> print(name + f"{k:d}-fold 교차 검증 정확도: {accuracy_scores.mean() * 100:.2f} (+/- {accuracy_scores.std() * 200:.2f})")
>>>
>>> model.fit(train_xs, train_y)
>>> os.mkdir('OUTPUT/' + name + '-IMAGES')
>>> impute(target_xs, model, raster_info, outdir='OUTPUT/' + name + '-IMAGES', class_prob=True, certainty=True)
RF5-fold 교차 검증 정확도: 89.88 (+/- 7.39)
ET5-fold 교차 검증 정확도: 89.57 (+/- 7.73)
XGB5-fold 교차 검증 정확도: 90.08 (+/- 7.12)
LGBM5-fold 교차 검증 정확도: 90.26 (+/- 7.52)
OUTPUT 디렉토리에 가보면, 머신러닝 결과로 우리는 각 분류기명으로 생성된 폴더 안에 총 네 가지의 GeoTiff 파일들이 생성된 것을 확인할 수 있다.
그중 머신러닝 예측결과 (0 or 1)를 갖는 response.tif와 종 적합성에 관련한 생존확률(0 ~ 1) 값을 갖는 probability_1.0.tif을 보고 결과를 해석할 수 있다.
실제 response.tif 파일을 사진 편집기로 열어보면 검게 칠해져 있어 머신러닝 결과 '실제 종이 생존하는 영역이 존재하는가?'에 대해 궁금해질 수 있는데, 이는 collections.Counter 객체를 통해 간단하게 확인할 수 있다.
>>> from collections import Counter
>>> Counter(distr_ex.flatten())
Counter({0: 54790, 1: 4010})
혹은 QGIS로 열람하여 벤더 렌더링에 의해 다음과 같이 생존여부 지도학습 결과를 쉽게 볼 수 있다.
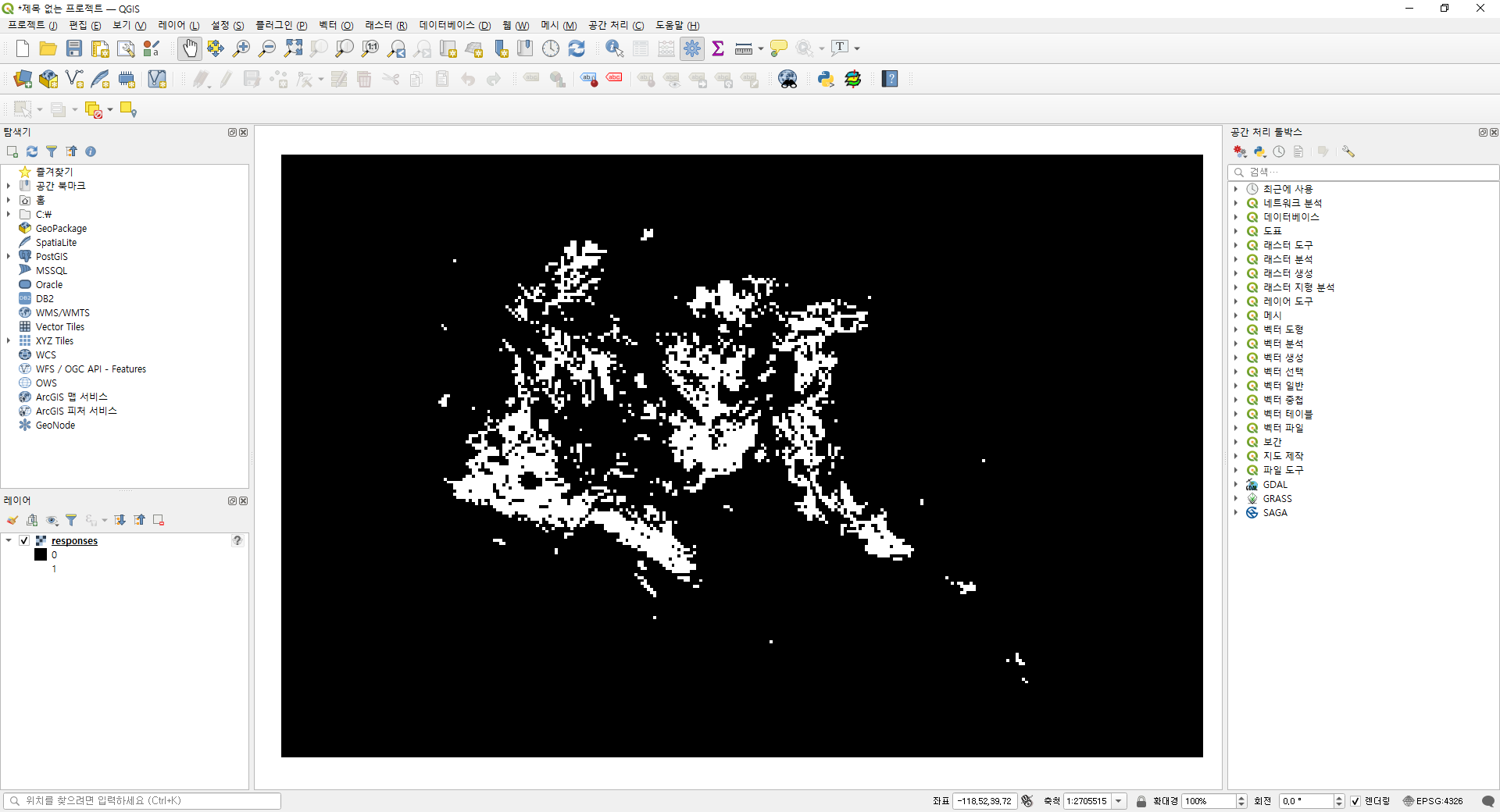
3. Visualization
위 데이터 분석 결과에 기반한 조슈아 트리 관련 의사결정을 내린다고 가정하면, 머신러닝 결과로 나온 종의 생존여부도 중요하지만 종 생존여부 가능성을 의미하는 확률값도 중요하다. 같은 생존지역이라고 할지라도 생존 가능성이 높은 지역보다 낮은 지역을 중심으로 하는 정책이 그 예가 될 것이다.
이 같은 경우 숫자(number)에 비교적 익숙하지 않은 의사결정자들이 좀 더 편하게 결과를 확인할 수 있도록 우리는 각 머신러닝 분류기에 따른 생존확률을 평균치를 가시화하할 수 있다.
from pylab import plt
def plotit(x, title, cmap="Blues"):
plt.imshow(x, cmap=cmap, interpolation='nearest')
plt.colorbar()
plt.title(title, fontweight = 'bold')
import rasterio
distr_rf = rasterio.open("outputs/rf-images/probability_1.0.tif").read(1)
distr_et = rasterio.open("outputs/et-images/probability_1.0.tif").read(1)
distr_xgb = rasterio.open("outputs/xgb-images/probability_1.0.tif").read(1)
distr_lgbm = rasterio.open("outputs/lgbm-images/probability_1.0.tif").read(1)
distr_averaged = (distr_rf + distr_et + distr_xgb + distr_lgbm)/4
plotit(distr_averaged, "Joshua Tree Range, averaged", cmap="Greens")
(참고) rasterio의 .read(1)은 첫 번째 밴드를 읽는다는 것을 의미한다. QGIS에서 probability_1.0.tif의 정보를 확인하면 밴드 개수가 한 개로 유일함을 확인할 수 있다.
Summary
pyimpute패키지를 사용하여 지리정보시스템을 활용한 머신러닝을 수행한다.- k-fold 교차검증 기법을 사용하여 특정 데이터에 의한 과적합 문제를 해결한다.
sklearn.model_selection을 통해 여러가지 머신러닝 분류기 모델에 의한 결과를 정확도를 지표로 모델을 평가한다.- 결과물을 시각화하여 의사결정 과정에 이용한다.
References
- Daniel, F. (2021). Pthon-species-distribution-modeling [Python]. link
- Matthew, P. (2020). pyimpute [Python]. link
- GGRS. (2022, January 21). QGIS와 Python을 이용한 종 분포 모델링(SDM) [Video], Youtube. link
- WorldClim. (2020). Bioclimatic variables. link
- Rasterio Offical Docs. Reading Datasets. link
- itholic. (2018). 파이썬 특정 폴더(디렉토리) 파일 리스트 가져오기. link
- programmerpsy. (2021). 레스터 데이터 분석. link






댓글 영역